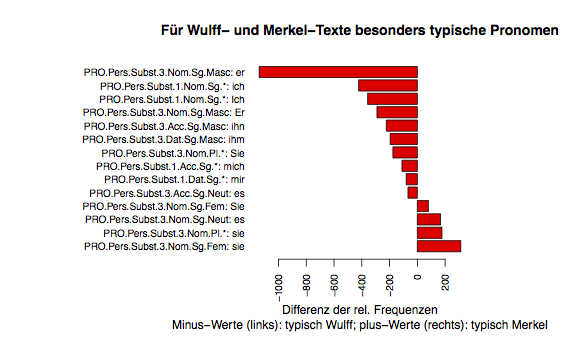
Ich bin etwas stolz auf die folgende Grafik:
Allerdings nicht, weil sie besonders schön aussehen würde oder weil der Inhalt sehr überraschend wäre, sondern weil sie methodisch elegant erstellt wurde. Nämlich in R unter direktem Zugriff auf die Corpus Workbench. Allerdings ist der Weg dahin doch etwas umständlich.
R mausert sich in der Korpuslinguistik zur Standardsoftware, um statistische Berechnungen anzustellen. Die Corpus Workbench ist ein verbreitetes Korpusmanagment- und Abfragesystem mit der mächtigen Query-Sprache CQP. Bisher fehlte ein Bindeglied: Die Möglichkeit, direkt in R auf die CWB zuzugreifen. Seit Februar diesen Jahres gibt es von Bernard Desgraupes und Sylvain Loiseau jedoch eine Lösung: Das rcqp-Paket.
Installation
Update 25. Juni 2012: Zumindest für Unix-Systeme sollte die Installation nun ganz einfach sein, da rcqp als Plug-in zu R vorkompiliert in CRAN-Repository liegt.
Momentan scheint es nur für Mac OS X und Windows nur die Möglichkeit zu geben, rcqp selber auf dem System zu kompilieren. Dafür sind einige Entwickler-Tools notwendig wie GCC, GNU make, GNU bison und flex etc., was auf Linux- und Mac OS X-Systemen normalerweise bereits installiert ist.
Zudem sind zwei weitere Bibliotheken notwendig: Die PCRE– und die Glib-Libraries, die wieder abhängig von weiteren Paketen sind. Wer da nachinstallieren muss, ist ggf. eine Weile damit beschäftigt.
Dann jedoch holt man sich die aktuellste Version von rcqp über SVN:
$ svn checkout svn://scm.r-forge.r-project.org/svnroot/rcwb/pkg/rcqp
Wechselt dann ins Verzeichnis pkg/rcqp/ und lässt R dann alles kompilieren und installieren:
$ cd rcwb/pkg/rcqp $ sudo R CMD INSTALL rcqp
Achtung: In der aktuellen INSTALL-Info steht ein falscher Befehl („install“ klein geschrieben statt groß). Doch dann ist, wenn alles gut läuft, das Paket installieren und man kann in R damit arbeiten.
Verwendung
Die Dokumentation führt Schritt für Schritt durch eine typische Korpusrecherche mit rcqp und erläutert alle verfügbaren Funktionen.
Der Code, um die Grafik oben zu erzeugen, sieht folgendermaßen aus: Erstmal muss die Bibliothek geladen werden und in der zweiten Zeile werden die verfügbaren Korpora ausgegeben:
library(rcqp); cqi_list_corpora(); [1] "MERKELKORPUS" "WULFFKORPUS"
Ich wähle nun das Korpus „MERKELKORPUS“ und mache darin mit subcorpus eine Abfrage, bei der ich die CQP-Syntax verwenden kann. Hier wird die Annotation des RFTaggers nach Personalpronomen durchsucht. Mit print kann man sich KWIC-Zeilen ausgeben lassen.
merkel <- corpus("MERKELKORPUS");
pMerkel <- subcorpus(merkel, '[rfpos="PRO.Pers.+"]');
print(pMerkel, from=1, to=10);
17 elche Politiker haben << Sie >> in Ihrem Leben am me
27 ndruckt ? Darauf habe << ich >> keine eindeutige Ant
34 wort . Adenauer halte << ich >> für einen großen Sta
41 n Staatsmann , obwohl << er >> mich nicht mochte .
42 taatsmann , obwohl er << mich >> nicht mochte . Mit B
50 kanzler Brandt konnte << ich >> gut . Er hat mich se
53 andt konnte ich gut . << Er >> hat mich sehr unters
66 en in den Osten . Wen << ich >> sehr schätze , ist A
74 ndeskanzler Schmidt . << Er >> ist geradlinig und s
81 dlinig und sagt , was << er >> denkt . Woher kommt
Die cqp_ftable-Funktion erzeugt eine schöne Distributionstabelle um zu sehen, wie sich die Treffer verteilen, in unserem Fall, auf welche Subklassen der Personalpronomen und welche Wortformen:
dMerkel <- cqp_ftable(pMerkel, "match", "rfpos", "match", "word"); dMerkel;
Ausgabe von cqp_ftable:
match.rfpos match.word freq 1 PRO.Pers.Subst.3.Nom.Sg.Neut es 10733 2 PRO.Pers.Subst.3.Nom.Sg.Masc er 8859 3 PRO.Pers.Subst.3.Nom.Sg.Fem sie 5126 4 PRO.Pers.Subst.3.Nom.Sg.Neut Es 4407 5 PRO.Pers.Subst.1.Nom.Pl.* wir 3566 6 PRO.Pers.Subst.3.Nom.Pl.* sie 3480 7 PRO.Pers.Subst.3.Nom.Pl.* Sie 3165 [...]
Genau das Gleiche machen wir mit dem Wulff-Korpus und vereinigen die beiden Tabellen:
wulff <- corpus("WULFFKORPUS");
pWulff <- subcorpus(wulff, '[rfpos="PRO.Pers.+"]');
dWulff <- cqp_ftable(pWulff, "match", "rfpos", "match", "word");
data <- merge(dMerkel, dWulff, by=c("match.rfpos", "match.word"));
colnames(data) <- c("rfpos", "word", "freqMerkel", "freqWulff");
data enthält nun folgende Tabelle:
data; rfpos word freqMerkel freqWulff 1 PRO.Pers.Subst.1.Acc.Pl.* uns 433 457 2 PRO.Pers.Subst.1.Acc.Pl.* Uns 1 2 3 PRO.Pers.Subst.1.Acc.Pl.* UNS 7 1 4 PRO.Pers.Subst.1.Acc.Sg.* mich 420 676 5 PRO.Pers.Subst.1.Acc.Sg.* Mich 18 40 6 PRO.Pers.Subst.1.Dat.Pl.* uns 600 654 7 PRO.Pers.Subst.1.Dat.Pl.* Uns 16 35 [...]
Mit der Funktion summary kann man wichtige Informationen zum Korpus ausgeben, unter anderem steht in der ersten Zeile die Korpusgröße in Anzahl Tokens:
summary(merkel); MERKELKORPUS Number or tokens in the corpus: 2584948
Diese Information verwenden wir, um relative Frequenzen zu berechnen. Gesondert für die Merkel- und die Wulff-Frequenzen fügen wir je eine Spalte mit den relativen Werten ein und eine weitere, die die Differenz enthält:
data$relMerkel <- data$freqMerkel/2584948*1000000; data$relWulff <- data$freqWulff/2474827*1000000; data$diff <- data$relMerkel - data$relWulff;
Diese Tabelle sortieren wir nun absteigend nach dem Differenzwert und erstellen dann eine Matrix-Tabelle, die alle Zeilen enthält, deren Differenzwerte > 50 oder < -50 sind:
newdata <- data[order(-data$diff),]; big50 <- newdata[newdata$diff > 50,]; little50 <- newdata[newdata$diff < - 50,]; biglittle50 <- rbind(big50, little50); biglittle50data <- as.matrix(data.frame(biglittle50$diff, row.names=paste(biglittle50$rfpos, biglittle50$word, sep=": ")));
Daraus ergibt sich folgende Tabelle:
biglittle50data;
biglittle50.diff
PRO.Pers.Subst.3.Nom.Sg.Fem: sie 313.00289
PRO.Pers.Subst.3.Nom.Pl.*: sie 177.28472
PRO.Pers.Subst.3.Nom.Sg.Neut: es 167.18947
PRO.Pers.Subst.3.Nom.Sg.Fem: Sie 79.63236
PRO.Pers.Subst.3.Acc.Sg.Neut: es -67.57275
PRO.Pers.Subst.1.Dat.Sg.*: mir -80.11974
PRO.Pers.Subst.1.Acc.Sg.*: mich -110.67132
PRO.Pers.Subst.3.Nom.Pl.*: Sie -176.10593
PRO.Pers.Subst.3.Dat.Sg.Masc: ihm -195.44418
PRO.Pers.Subst.3.Acc.Sg.Masc: ihn -223.50883
PRO.Pers.Subst.3.Nom.Sg.Masc: Er -290.77445
PRO.Pers.Subst.1.Nom.Sg.*: Ich -357.91871
PRO.Pers.Subst.1.Nom.Sg.*: ich -423.05258
PRO.Pers.Subst.3.Nom.Sg.Masc: er -1138.82748
Daraus können wir nun die Grafik, einen barplot, erstellen und in eine PDF-Datei schreiben:
pdf("PronomenDiff.pdf", width=8, height=5);
par(mar=c(7, 18, 4.1, 8));
par(las=2);
par(cex.axis=0.8);
barplot(t(biglittle50data), main="Für Wulff- und Merkel-Texte
besonders typische Pronomen", horiz=T, xlab="Differenz der
rel. Frequenzen", sub="Minus-Werte (links): typisch Wulff;
plus-Werte (rechts): typisch Merkel", col="red");
dev.off();
Fazit
Leute, die sich gut mit R auskennen und mit Corpus Workbench-Korpora arbeiten, werden Freude an diesem Modul haben. Denn ansonsten muss man immer zwischen den beiden Tools hin und her wechseln und Zwischenergebnisse exportieren und importieren. Allerdings ist die Installation relativ aufwändig und nur für Erfahrene geeignet. Im Prinzip sollte das Modul auch unter Windows funktionieren, was ich jedoch nicht ausprobierte.
Eine Alternative bietet z.B. Roland Schäfer mit seinem online Koka-Konvertierungstool, das CWB-KWICs (und auch IDS DeReKo/COSMAS II und DWDS-KWICs) in Excel-Tabellen konvertieren kann.